この記事はStan Advent Calendar 2019 18日目の内容になります。
ベイズ流臨床試験デザイン
みなさん今年もStanアドカレ楽しんでいますか。アドカレで毎日Stanやベイズ推定のtipsが読める12月はとても幸せですね。
今日はStanを使って臨床試験のデザインや解析を行う方法の一部を紹介します。
近年、臨床試験の効率化等の観点から、試験の結果を逐次的にモニタリングし、経過に応じて柔軟な意思決定を行いやすいベイズ流の臨床試験デザインの活用が期待されています1。
ベイズ流の臨床試験デザインには、第2相試験または3相で適用されるものが多く開発されており、予測確率に基づく有効性の逐次モニタリング、サンプルサイズ再設定、適応的ランダム化などがあります。それらに関しては、他の書籍や論文などでも紹介されています2。本日は個人的には日本語での紹介がやや少ないかな?と思う、第1相試験で薬剤処方用量を決める際に用いられている連続再評価法(Continual Reassessment Method: CRM)について取り上げます。
第1相試験
CRMは臨床試験の最初の相、第1相試験で適用されます。ここでの目的は、試験薬の最大耐用量(mamixmum tolerated dose: MTD)を見積もることです。MTDとは、重篤な副作用を引き起こすことなく投与できる薬物または治療の最大の用量を指します。つまり、安全なレベルでどこまで処方量を引きあげて良いかというのを探ることが第1相試験での主要な目的となるのです。
CRMは主には、薬剤の処方用量の探索に用いられていますが、rTMSやtDCSあるいはニューロフィードバックのような、一定の刺激呈示を1回の処方とするような精神疾患の治療的介入の最適回数の探索にも使えるようです。アプリを用いた心理学的な介入(プロンプトの呈示)なども最適回数を探索するのに使えそうです。
推定するモデル
いくつかの亜型が存在しますが、基本的には、用量と毒性発生確率の用量反応曲線(関数)を仮定し、その曲線を規程するパラメータを推定します。
\(x_i\)を標準化処方量とし、特定の処方量における用量制限毒性(dose limiting toxicity:DLT)の確率を、\(F(x_i,\theta)\)といった平滑化関数を用いて推定します。ここで、\(\theta\)はパラメータベクトルです。関数\(F\)の設定を少しずつ変えた亜型が存在します。
\(Y_i\)が\(\{0,1\}\)の2値をとるランダム変数で、患者\(Patient_i\)におけるDLTの有無を表しているとします。ある処方量\(x_i\)を投与された患者における毒性の推定確率が\(F(x_i、\theta)\)であるから、患者\(patient_i\)の尤度は、
\[F(x_i,\theta)^{Y_i}(1-F(x_i,\theta))^{1-Y_i}\tag{1}\]
となり、 複数の患者\(J\)を評価した後の尤度は、
\[L_j(\theta)=\prod_{i=1}^{J} \left \{ F(x_i,\theta)\right \}^{Y_i}\left \{1-F(x_i,\theta)\right \}^{1-Y_i}\tag{2}\]
となります。
trialパッケージのstan_crm関数で指定可能なCRMの亜型は以下の4つです。
\[F(x_i,\beta)=x_i^{exp \beta}\tag{3}\]
\[F(x_i,\beta)=1/(1+exp(-a_0-exp(\beta)x_i))\tag{4}\]
\[F(x_i,\beta)=1/(1+exp(-a_0-\beta x_i))\tag{5}\]
\[F(x_i,\beta)=1/(1+exp(-a-exp(\beta)x_i))\tag{6}\]
- 式 (3)では、\(\beta\)に正規分布を仮定
- 式 (4)では、\(a_0\)固定母数, \(\beta\)は正規分布
- 式 (5)では、\(a_0\)固定母数, \(\beta\)はガンマ分布
- 式 (6)では、\(a\)が自由母数になり、\(a\)も\(\beta\)も正規分布
です。
モデルのイメージをつかむために、関数を可視化してみます。ここで式4の1パラメータのロジスティックモデルで書いてみると、横軸に処方量、縦軸に毒性発生確率をとって用量反応曲線を描くことができます。
crm.f<-function(beta){
d<-seq(0,5,length=1000)
p=NULL
for(i in d){
p<-c(p,exp(-3+beta*i)/(1+exp(-3+beta*i)))
}
df<-data.frame(probability=p,std.dose=d)
require(ggplot2)
qplot(y=probability,x=std.dose,data=df)+geom_line()+theme_bw()+ylim(0,1)
}
library(purrr)
library(gridExtra)
list(10,2,1)%>% map(crm.f)-> p
grid.arrange(grobs = p, ncol =1)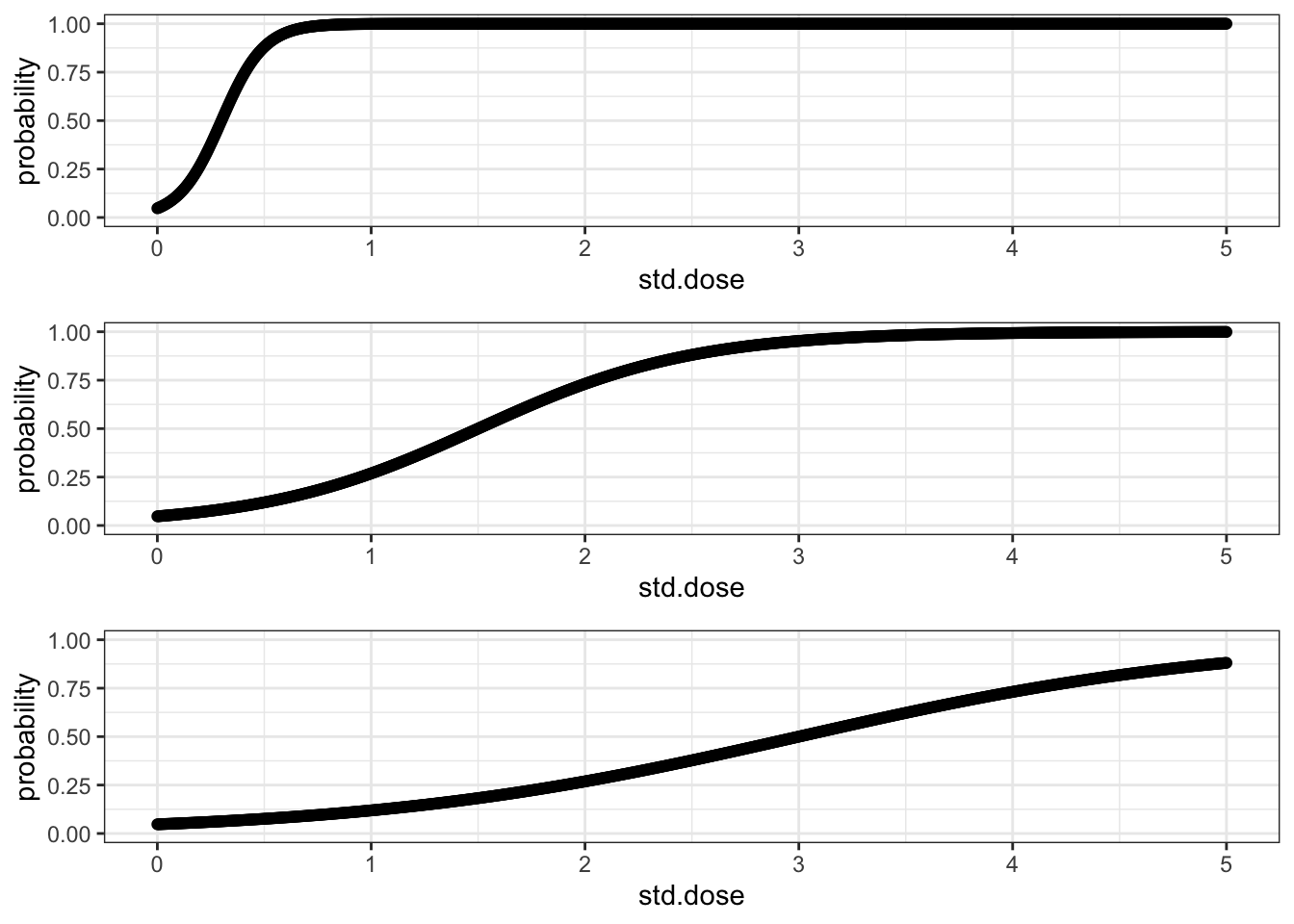
\(a_0\)は固定母数なので、3つのプロットは\(\beta\)の値が異なるのみです。 \(\beta\)は用量反応曲線の傾きを反映するパラメータになっています。今回の場合は上、中、下段の順で\(\beta\)の値(傾き)が小さく、上段の図では用量が低い段階で毒性の発現確率が高く、中段では中程度の用量で発現確率が高くなり、下段では高用量で毒性の発現確率が高くなる、といったかたちで、用量反応関係を記述することが可能となります。この用量反応曲線の型を規定する\(\beta\)をベイズ推定で求めようというのがCRMの基本的な発想となります。
また、CRMではこの割合を超えて毒性が発生して欲しくないという、標的となる毒性発現確率を定めます。上の図で標的となる毒性発現確率を0.25と設定した場合に中段の図では処方量がおよそ1のあたりで毒性発現確率が0.25となっており、MTDは1付近の(1より小さい)値となります。
trialr パッケージ
trialrパッケージに、上記の4つのCRMモデルをStanで実行可能な関数が実装されています。trialr自体の使い方は非常に簡単です。
5段階の処方用量があり、各用量の毒性発現確率の見積もり(skeleton)を、\(\{0.05,0.15,0.25,0.4,0.6\}\)とし、標的となる毒性発現確率は0.25としたとします(つまり毒性発現確率が25%を超えない用量が望ましい)。
library(trialr)
target<-0.25
skeleton<-c(0.05,0.15,0.25,0.4,0.6)この設定のもとで、次のデータを得たとします。 全部で6名の患者のうち2名、用量水準2で投与したのち、次の2名には水準3で投与、次の2名には水準4で投与し、水準4の時に2名が毒性を発現した、というデータです。
data<-data.frame(Patient=1:6,
Cohort =rep(1:3,each=2),
Dose_level=rep(2:4,each=2),
DLT = c(0,0,0,0,1,1))
data## Patient Cohort Dose_level DLT
## 1 1 1 2 0
## 2 2 1 2 0
## 3 3 2 3 0
## 4 4 2 3 0
## 5 5 3 4 1
## 6 6 3 4 1trialrのstan_crm関数では、データの指定の仕方が独特で上のデータは下記のように表現されます。
outcomes <- '2NN 3NN 4TT'2NNとは用量水準2で投与した人が二人とも毒性なし(NN)であることを表しています。もし、1人目に毒性があって、2人目に毒性がなければ2TNとなります。
3NNは同様に、用量水準3で投与した人が二人とも毒性なしなのでNN、4TTは用量水準4で投与した2人とも毒性発現ありなのでTTとなります。
データの準備ができたら、あとはstan_crmに適切な引数指定すればOKです。
式(4),(5),(6)のロジスティックモデルはそれぞれ、下記のように指定します。 各モデルの定数(\(a_0\))や事前分布のパラメータは、Wheeler (2019) が参考になります。
- 式4: 1パラメータロジスティックモデル, 正規分布
model1<-stan_crm(outcomes,
skeleton = skeleton,
target = target,
model = 'logistic',
a0=3, beta_mean=0, beta_sd = sqrt(1.34))- 式5: 1パラメータロジスティックモデル, ガンマ分布
model2<-stan_crm(outcomes,
skeleton = skeleton,
target = target,
model = 'logistic_gamma',
a0=3, beta_shape=1, beta_inverse_scale = 1)- 式6: 2パラメータロジスティックモデル, 正規分布
model3<-stan_crm(outcomes,
skeleton = skeleton,
target = target,
model = 'logistic2',
alpha_mean =0, alpha_sd=1,
beta_mean=0,beta_sd=1)推定結果のサマリーが下記で示されます。各処方用量における、毒性発現確率(prob_tox)を、それぞれのモデルでプロットしてみます。
three_model<-data.frame(prob_tox=c(model1$prob_tox,model2$prob_tox,model3$prob_tox),
model=rep(1:3,each=5),
dose=rep(1:5,3))
ggplot(three_model,
aes(x=dose,y=prob_tox,color=as.factor(model)))+
geom_line()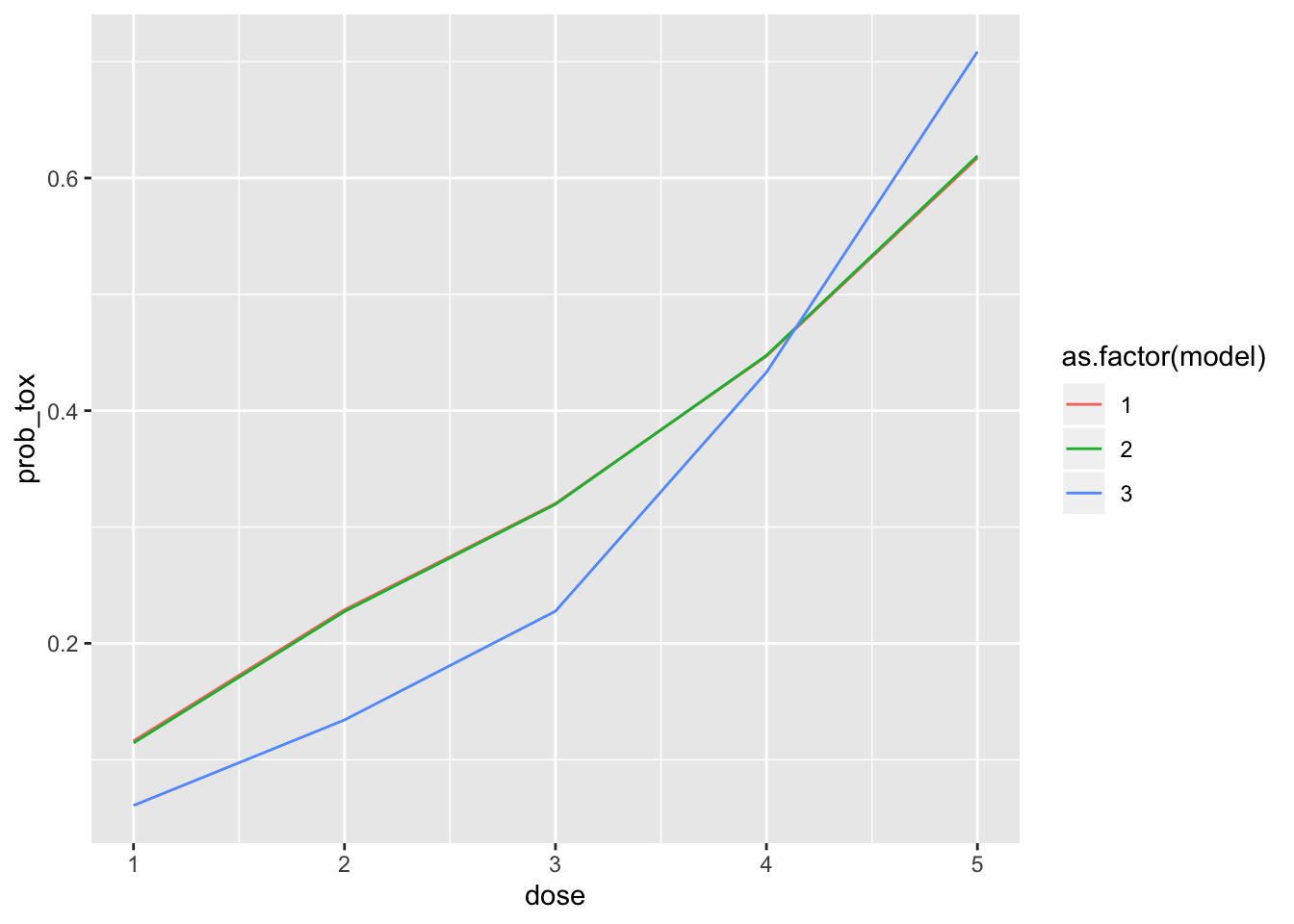
今回は標的となる毒性発現確率が0.25で、model1と2はMTDが2と3の間、model3では3を少し越えた値となりました。なので、model1と2ではMTDは2、model3ではMTDは3という判断となります。
stan_crmを実行したオブジェクトは、stanやbrmsと同様にtidybayesパッケージの関数が適用可能です。これはとても気持ちいいことです。tidybayesの使いかたはこちら。サマリーの推定値だけではベイズ感が乏しいので、事後分布をプロットしてみましょう。
library(tidyverse)
library(tidybayes)
model1 %>%
spread_draws(prob_tox[Dose]) %>%
ggplot(aes(x = Dose, y = prob_tox)) +
stat_interval(.width = c(.5, .8, .95)) +
scale_color_brewer() +
labs(y = 'Prob(DLT)', title = '各用量の毒性発現確率の事後分布')+
theme_gray (base_family = "HiraKakuPro-W3")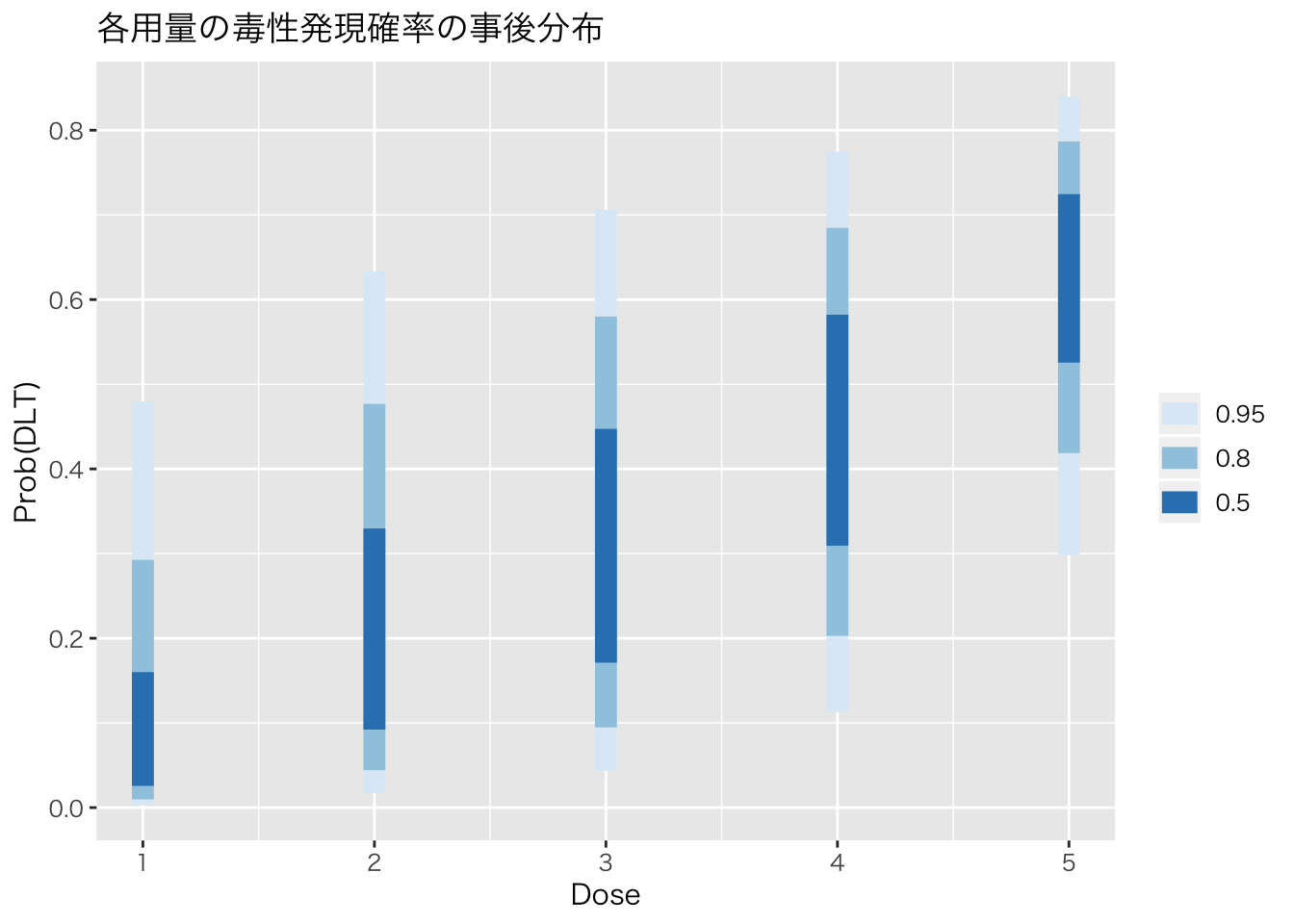
中で走らせてるstan コード
trialrパッケージのgithubのサイトに、各モデルのstan codeがあるので、それをみてみましょう。
下記は[CrmOneParamLogisticNormalPrior.stan]というファイルに書かれた、正規分布をパラメータの事前分布にした、1パラメータのロジスティックモデルのstanコードです。
stanのfunctionsで関数\(F(x_i, \beta) = exp\{a_0 + exp(beta) x_i\} / (1 +exp\{a_0 + exp(beta) x_i\})\)を定義しています。
\(x_1\) : skeleton (事前の各用量の毒性発現確率)
\(a_0\) : 切片, 定数
\(\beta\) : 傾き, 推定するパラメータ
\(\beta \sim N(\beta_{mean}, \beta_{sd})\)
betaにexpをかましているのは、単調増加の用量反応曲線が正値をとるようにするためとのことです。またこの関数ではweightsで重みも加えられるようになっています。
LOGIT1PLNORM.crm<-
'
functions {
real log_joint_pdf(int num_patients, int[] tox, int[] doses, real[] weights,
real[] codified_doses, real a0, real beta) {
real p;
p = 0;
for(j in 1:num_patients) {
real prob_tox;
real p_j;
prob_tox = inv_logit(a0 + exp(beta) * codified_doses[doses[j]]);
p_j = (weights[j] * prob_tox)^tox[j] *
(1 - weights[j] * prob_tox)^(1 - tox[j]);
p += log(p_j);
}
return p;
}
}
data {
// betaの事前分布(正規分布)の平均と標準偏差
real beta_mean;
real<lower=0> beta_sd;
// 用量の種類数
int<lower=1> num_doses;
// skeleton (各処方用量における毒性発現確率の事前の見積もり). skeletonは単調増加で指定.
real<lower=0, upper = 1> skeleton[num_doses];
// 切片定数: 3が推奨
real a0;
// 対象者数
int<lower=0> num_patients;
// 対象者ごとの毒性の有無(2値)
int<lower=0, upper=1> tox[num_patients];
// 用量水準
int<lower=1, upper=num_doses> doses[num_patients];
// 重み0から1 TITE-CRMで使用
real weights[num_patients];
}
transformed data {
// Codified dosesに、skeletonの値を用量反応関係の値となるよう変換し格納
real codified_doses[num_doses];
for(i in 1:num_doses) {
codified_doses[i] = (logit(skeleton[i]) - a0) / exp(beta_mean);
}
}
parameters {
// 推定するパラメータはbetaだけ
real beta;
}
transformed parameters {
// 推定したbetaを用いて、各処方用量における毒性発現確率prob_toxの事後分布を推定
real<lower=0, upper=1> prob_tox[num_doses];
for(i in 1:num_doses) {
prob_tox[i] = inv_logit(a0 + exp(beta) * codified_doses[i]);
}
}
model { // betaはN(beta_mean, beta_sd)の正規分布
target += normal_lpdf(beta | beta_mean, beta_sd);
// functionブロックで定義したlog_joint_pdfを使用
target += log_joint_pdf(num_patients, tox, doses, weights, codified_doses, a0, beta);
}
generated quantities { // 対数尤度の計算
vector[num_patients] log_lik;
for (j in 1:num_patients) {
real p_j;
p_j = inv_logit(a0 + exp(beta) * codified_doses[doses[j]]);
log_lik[j] = log((weights[j] * p_j)^tox[j] *
(1 - weights[j] * p_j)^(1 - tox[j]));
}
}
'library(rstan)
LOGIT1PLNORM.crm<-stan_model(model_code=LOGIT1PLNORM.crm)上のstanコードを実行してみるために、dataブロックに合わせてデータを加工のします。ここではちょっとずるして、model1のstan_crmオブジェクトに格納されているデータを援用します。中身はstanで走らせるために、list型になっています。
model1$dat## $num_doses
## [1] 5
##
## $skeleton
## [1] 0.05 0.15 0.25 0.40 0.60
##
## $target
## [1] 0.25
##
## $a0
## [1] 3
##
## $alpha_mean
## NULL
##
## $alpha_sd
## NULL
##
## $beta_mean
## [1] 0
##
## $beta_sd
## [1] 1.157584
##
## $beta_shape
## NULL
##
## $beta_inverse_scale
## NULL
##
## $num_patients
## [1] 6
##
## $doses
## [1] 2 2 3 3 4 4
##
## $tox
## [1] 0 0 0 0 1 1
##
## $weights
## [1] 1 1 1 1 1 1
##
## attr(,"class")
## [1] "crm_params" "list"あたりまえですが、先ほどstan_crm関数で走らせたのと、同じ結果が出ます。
library(rstan)
fit<-rstan::sampling(LOGIT1PLNORM.crm,data=model1$dat)fit## Inference for Stan model: 1796da600fc84aff506dab46fe3991e0.
## 4 chains, each with iter=2000; warmup=1000; thin=1;
## post-warmup draws per chain=1000, total post-warmup draws=4000.
##
## mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
## beta -0.08 0.01 0.28 -0.68 -0.25 -0.06 0.10 0.40 1089 1
## prob_tox[1] 0.12 0.00 0.13 0.00 0.03 0.07 0.16 0.50 961 1
## prob_tox[2] 0.23 0.01 0.17 0.02 0.09 0.19 0.33 0.64 1012 1
## prob_tox[3] 0.32 0.01 0.19 0.04 0.18 0.30 0.45 0.72 1075 1
## prob_tox[4] 0.45 0.01 0.18 0.11 0.31 0.45 0.58 0.78 1170 1
## prob_tox[5] 0.62 0.00 0.14 0.30 0.53 0.64 0.73 0.84 1297 1
## log_lik[1] -0.30 0.01 0.28 -1.04 -0.41 -0.21 -0.10 -0.02 971 1
## log_lik[2] -0.30 0.01 0.28 -1.04 -0.41 -0.21 -0.10 -0.02 971 1
## log_lik[3] -0.44 0.01 0.33 -1.26 -0.60 -0.36 -0.19 -0.04 992 1
## log_lik[4] -0.44 0.01 0.33 -1.26 -0.60 -0.36 -0.19 -0.04 992 1
## log_lik[5] -0.91 0.01 0.52 -2.19 -1.16 -0.79 -0.54 -0.25 1402 1
## log_lik[6] -0.91 0.01 0.52 -2.19 -1.16 -0.79 -0.54 -0.25 1402 1
## lp__ -4.38 0.03 0.83 -6.68 -4.59 -4.07 -3.85 -3.79 1049 1
##
## Samples were drawn using NUTS(diag_e) at Wed Dec 18 18:11:18 2019.
## For each parameter, n_eff is a crude measure of effective sample size,
## and Rhat is the potential scale reduction factor on split chains (at
## convergence, Rhat=1).fit %>%
spread_draws(prob_tox[Dose]) %>%
ggplot(aes(x = Dose, y = prob_tox)) +
stat_interval(.width = c(.5, .8, .95)) +
scale_color_brewer() +
labs(y = 'Prob(DLT)', title = '各用量の毒性発現確率の事後分布')+
theme_gray (base_family = "HiraKakuPro-W3")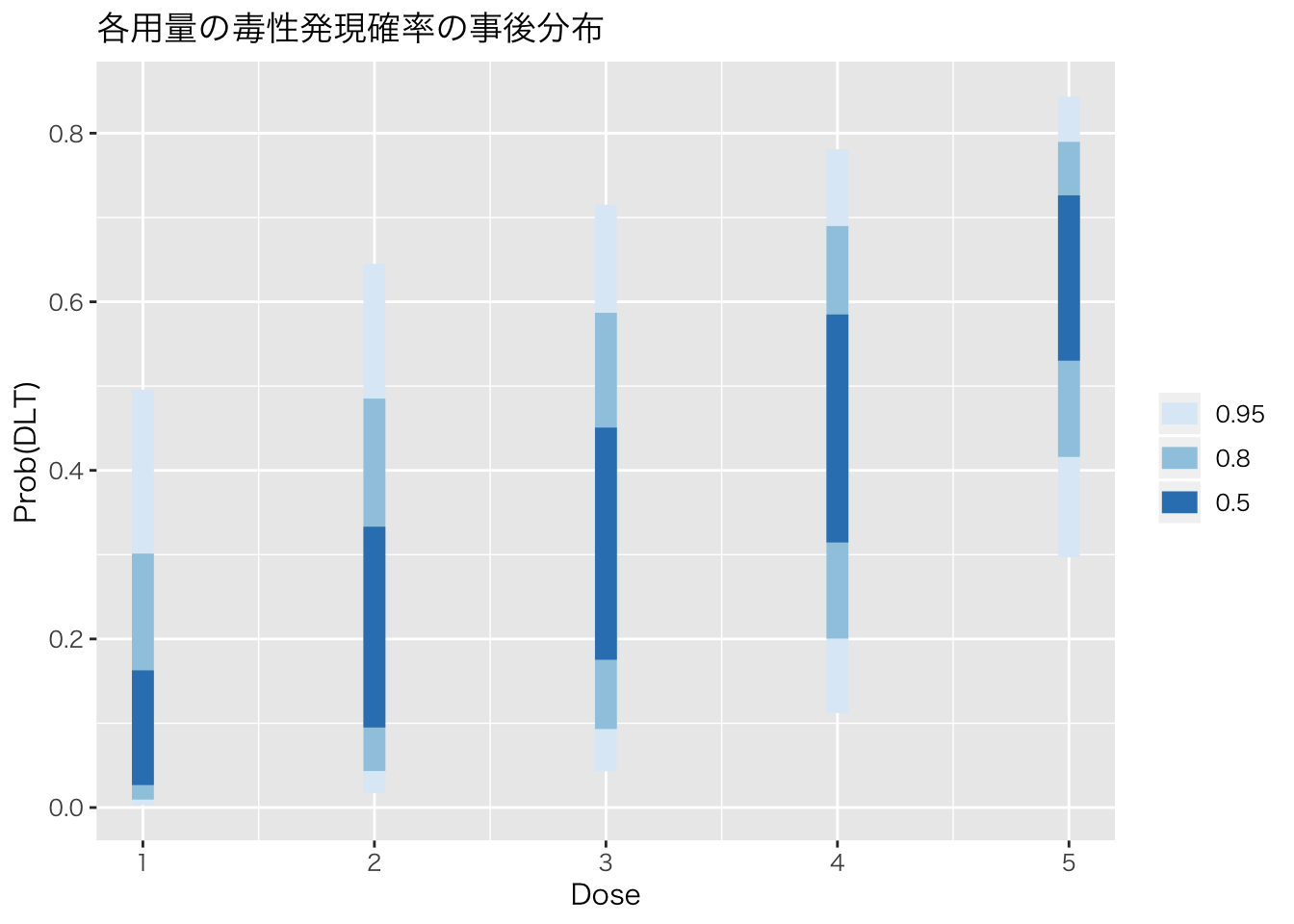
githubのサイトには、trialパッケージに含まれるCRMの各種モデルのstanコードが収容されているので、関心のあるかたはそれぞれのコードを眺めてみると面白いと思いますし、結果的に、trialパッケージでいいよねってなりますのでおすすめです。独自の生成量を追加したい時などは、元のstanコードが役にたちます。
今日はCRMも基本的なモデル(1母数、2母数ロジスティクモデル)だけをみましたが、trialrではさらに発展的なモデルも扱うことができます。
Enjoy!!
Session info
sessionInfo()## R version 3.6.1 (2019-07-05)
## Platform: x86_64-apple-darwin15.6.0 (64-bit)
## Running under: macOS High Sierra 10.13.3
##
## Matrix products: default
## BLAS: /Library/Frameworks/R.framework/Versions/3.6/Resources/lib/libRblas.0.dylib
## LAPACK: /Library/Frameworks/R.framework/Versions/3.6/Resources/lib/libRlapack.dylib
##
## locale:
## [1] ja_JP.UTF-8/ja_JP.UTF-8/ja_JP.UTF-8/C/ja_JP.UTF-8/ja_JP.UTF-8
##
## attached base packages:
## [1] stats graphics grDevices utils datasets methods base
##
## other attached packages:
## [1] tidybayes_1.1.0 forcats_0.4.0 stringr_1.4.0 dplyr_0.8.3
## [5] readr_1.3.1 tidyr_1.0.0.9000 tibble_2.1.3 tidyverse_1.3.0
## [9] trialr_0.1.2 Rcpp_1.0.3 gridExtra_2.3 purrr_0.3.3
## [13] knitr_1.26 rstan_2.19.2 ggplot2_3.2.1 StanHeaders_2.19.0
##
## loaded via a namespace (and not attached):
## [1] httr_1.4.1 jsonlite_1.6
## [3] binom_1.1-1 modelr_0.1.5
## [5] gtools_3.8.1 assertthat_0.2.1
## [7] stats4_3.6.1 cellranger_1.1.0
## [9] ggstance_0.3.3 yaml_2.2.0
## [11] pillar_1.4.2 backports_1.1.5
## [13] lattice_0.20-38 glue_1.3.1
## [15] arrayhelpers_1.0-20160527 digest_0.6.23
## [17] RColorBrewer_1.1-2 rvest_0.3.5
## [19] colorspace_1.4-1 htmltools_0.4.0
## [21] plyr_1.8.5 pkgconfig_2.0.3
## [23] broom_0.5.2 svUnit_0.7-12
## [25] haven_2.2.0 bookdown_0.15
## [27] scales_1.1.0 processx_3.4.1
## [29] generics_0.0.2 farver_2.0.1
## [31] ellipsis_0.3.0 withr_2.1.2
## [33] lazyeval_0.2.2 cli_2.0.0
## [35] readxl_1.3.1 magrittr_1.5
## [37] crayon_1.3.4 evaluate_0.14
## [39] ps_1.3.0 fs_1.3.1
## [41] fansi_0.4.0 nlme_3.1-142
## [43] xml2_1.2.2 pkgbuild_1.0.6
## [45] blogdown_0.17 tools_3.6.1
## [47] loo_2.1.0 prettyunits_1.0.2
## [49] hms_0.5.2 lifecycle_0.1.0
## [51] matrixStats_0.55.0 reprex_0.3.0
## [53] munsell_0.5.0 callr_3.4.0
## [55] compiler_3.6.1 rlang_0.4.2
## [57] grid_3.6.1 rstudioapi_0.10
## [59] labeling_0.3 rmarkdown_1.18
## [61] gtable_0.3.0 codetools_0.2-16
## [63] inline_0.3.15 DBI_1.0.0
## [65] R6_2.4.1 rstantools_2.0.0
## [67] lubridate_1.7.4 stringi_1.4.3
## [69] parallel_3.6.1 vctrs_0.2.0.9007
## [71] dbplyr_1.4.2 tidyselect_0.2.5
## [73] xfun_0.11 coda_0.19-3例えば、 FDAの適応的デザインのガイドライン↩
例えば、 手良向 (2017)、 手良向・大門(訳)(2014)、 竹林由武 (2018)などがあります。↩