## Warning: パッケージ 'knitr' はバージョン 3.5.2 の R の下で造られました今年も始まりましたね。Stanアドカレ!! 3年連続のエントリーとなりました。本記事は、Stan adventcalender 2018 3日目の記事になります。
2日目のHojoさんの記事で登場したbayesplotパッケージのように、Stanでモデリングした結果を扱いやすくしてくれるパッケージがいくつか出されています。ここでは、tidybayesパッケージを紹介したいと思います。
tidybayesは,ミシガン大学情報学部の助教 Matthew Kayさんが開発したRのパッケージです。去年のアドカレで紹介したctmパッケージの開発もイケメンでしたが、これまたイケメンです。ちなみに昨日担当のHojoさんもイケメン(*´Д`)ハァハァです。

tidybayes: Bayesian analysis + tidy data + geoms
Rstanやbrmsなどでベイズ推定の推定結果を整然データ化して、dplyrの関数群やggplotで扱いやすくするぜ、パイプ演算子でホイホイ繋いで行こうぜ、これがtidybayesの基本的な発想です。
そもそも整然データとはなんぞや、という方はこちらの記事やこちらの本をご参照ください。
対応するパッケージ
tidybayesは下記の主要なベイズ推定パッケージのオブジェクトに対応しています。
- rstan, brms, rstanarm, runjags, rjags, jagsUI, coda::mcmc and coda::mcmc.list, MCMCglmm
ここでは、Stan advent calenderなので、Stanの推定結果を使っていきます。
分析に使用するデータとモデル
- 福島県の各市町村の平成24年の自殺者数と期待死亡数(福島県、自殺関連指標を計算するエクセルシートから整理)。なお、今回は例示のために適当な年度からデータをとってきており、平成24年を抽出したことに特別な意味はありません。また福島県のデータを扱っているのも、県で自殺数と期待死亡数をエクセルファイルで公表しているというデータのアクセスしやすさということで選んでいます。
データは以下のように.csvファイルから読み込みました。 59の市区町村のデータが縦に並んでいるデータです。
今回の分析に必要なデータやスクリプトはこちらから
library(knitr)
library(readxl)
dat<-read_excel("Sdat/H25fukushima_suicide.xlsx")
# d : 実自殺死亡数,exp_d:期待死亡数, area:行政区域
kable(dat)| ct_name | d | exp_d | area |
|---|---|---|---|
| 福島市 | 44 | 58.168141 | 県北 |
| 会津若松市 | 25 | 25.375612 | 会津 |
| 郡山市 | 60 | 25.375612 | 県中 |
| いわき市 | 68 | 67.907786 | いわき |
| 白河市 | 17 | 12.850420 | 県南 |
| 須賀川市 | 18 | 15.671975 | 県中 |
| 喜多方市 | 11 | 10.675036 | 会津 |
| 相馬市 | 10 | 7.348924 | 相双 |
| 二本松市 | 13 | 11.972770 | 県北 |
| 田村市 | 13 | 8.129224 | 県中 |
| 南相馬市 | 17 | 13.573328 | 相双 |
| 伊達市 | 17 | 13.312291 | 県北 |
| 本宮市 | 6 | 6.269023 | 県北 |
| 桑折町 | 6 | 2.605842 | 県北 |
| 国見町 | 1 | 2.070456 | 県北 |
| 川俣町 | 5 | 3.162235 | 県北 |
| 大玉村 | 1 | 1.723326 | 県北 |
| 鏡石町 | 5 | 2.527968 | 県中 |
| 天栄村 | 1 | 1.259279 | 県中 |
| 下郷町 | 1 | 1.334998 | 南会津 |
| 檜枝岐村 | 0 | 0.132899 | 南会津 |
| 只見町 | 1 | 1.027436 | 南会津 |
| 南会津町 | 9 | 3.653121 | 南会津 |
| 北塩原村 | 1 | 0.636893 | 会津 |
| 西会津町 | 2 | 1.531115 | 会津 |
| 磐梯町 | 1 | 0.766568 | 会津 |
| 猪苗代町 | 4 | 3.224526 | 会津 |
| 会津坂下町 | 1 | 3.509567 | 会津 |
| 湯川村 | 1 | 0.658855 | 会津 |
| 柳津町 | 2 | 0.805126 | 会津 |
| 三島町 | 0 | 0.407848 | 会津 |
| 金山町 | 1 | 0.531824 | 会津 |
| 昭和村 | 0 | 0.327850 | 会津 |
| 会津美里町 | 9 | 4.636978 | 会津 |
| 西郷村 | 5 | 3.950623 | 県南 |
| 泉崎村 | 0 | 1.352458 | 県南 |
| 中島村 | 0 | 1.013841 | 県南 |
| 矢吹町 | 3 | 3.685508 | 県南 |
| 棚倉町 | 4 | 2.969165 | 県南 |
| 矢祭町 | 2 | 1.300456 | 県南 |
| 塙町 | 1 | 2.008611 | 県南 |
| 鮫川村 | 1 | 0.796393 | 県南 |
| 石川町 | 6 | 3.618223 | 県中 |
| 玉川村 | 1 | 1.438024 | 県中 |
| 平田村 | 1 | 1.385329 | 県中 |
| 浅川町 | 3 | 1.381659 | 県中 |
| 古殿町 | 1 | 1.205864 | 県中 |
| 三春町 | 6 | 3.694287 | 県中 |
| 小野町 | 3 | 2.244206 | 県中 |
| 広野町 | 0 | 1.060691 | 相双 |
| 楢葉町 | 1 | 1.512063 | 相双 |
| 富岡町 | 3 | 2.926424 | 相双 |
| 川内村 | 1 | 0.590485 | 相双 |
| 大熊町 | 1 | 2.185874 | 相双 |
| 双葉町 | 2 | 1.308440 | 相双 |
| 浪江町 | 2 | 3.950420 | 相双 |
| 葛尾村 | 0 | 0.317573 | 相双 |
| 新地町 | 0 | 1.609630 | 相双 |
| 飯館村 | 2 | 1.262074 | 相双 |
市町村レベルの標準化死亡比推定モデル
標準化死亡比
標準化死亡比 (Standardized Mortality Ratio)は、地域間での死亡率を比較する指標としてよく用いられる指標です。
一般的には、当該地域の実際の死亡数\(d\)と、全国の年齢階級別死亡率と当該地域の死亡数かけて算出される期待死亡数\(exp_d\)の比をとって100かけた値,
\(\frac{d}{exp_d}\times 100\),
がSMRになります。
100であれば全国平均と同一、100以下であれば全国より当該地域の死亡率が低い、100以上であれば全国より当該地域の死亡率が高いことを示しています。
なお、SMRは人口規模の小さい地域では変動が大きいという欠点があり、それを補うために、階層ベイズモデルを用いた縮約統計量の利活用が推奨されています。
市区町村の標準化死亡比を推定するpoisson-gammaモデル
ここでは、上記の紺田先生のモデルに基づいて標準化死亡比を階層モデルを使って推定します。
- 地域\(i\)の自殺発生数: \(d_i\)
- 地域\(i\)の期待死亡数: \(exp\_d_i\)
- 地域\(i\)の潜在的な標準化死亡比: \(\theta_i\)
possison-gamma モデル
\[d_i \sim Poisson(exp\_d_i \theta_i)\]
\(\theta\)の事前分布に、ガンマ分布指定し、
\[ \theta_i \sim Gamma (\alpha, \beta) \]
ガンマ分布の形状パラメータ(\(\alpha\))と尺度パラメータ(\(\beta\))には、指数分布とガンマ分布を指定します。
\[\alpha \sim Exp (b_\alpha = 1), \beta \sim Gamma (a = 0.1, b_\beta = 1)\]
stan コード
このpoisson-gammaモデルの推定を行うためのstanコードは、以下のようになります。
data{
int r; // 地域数
int d[r]; //各地域の自殺発生数
real exp_d[r]; //各地域の期待死亡数
}
parameters{
real <lower=0> theta[r];
real <lower=0> alpha;
real <lower=0> beta;
}
model {
for(i in 1:r){
d[i] ~ poisson(exp_d[i]*theta[i]);
theta[i] ~ gamma(alpha,beta);
}
alpha ~ exponential(1);
beta ~ gamma(0.1,1);
}
generated quantities {
real RRmean;
real RRvar;
RRmean = alpha/beta; // 市区町村の自殺相対リスクの県平均
RRvar = alpha/(beta^2); // 市区町村の自殺相対リスクの分散
}
generated quantitiesブロックでは、gamma分布の性質利用して\(\alpha\)と\(\beta\)か県レベルの相対リスクの平均と分散を生成量として求めています。
推定の実行
library(rstan)
options(mc.cores = parallel::detectCores())
rstan_options(auto_write = TRUE)
modelGP<-stan_model("Sdat/HPGmodel.stan")
fit<-sampling(modelGP,data=list(d=dat$d,exp_d=dat$exp_d,r=nrow(dat)))ここからtidybayesの関数を紹介
spread_draw関数
tidybayesパッケージでは、ほぼ全ての場合において、ベイズ推定結果が格納されているオブジェクトをこのspread_draws関数に流すことから始まります。stanオブジェクトをspread_draws関数に落とすと下記のような整然データが得られます。
library(tidyverse)
library(tidybayes)
fit %>% spread_draws(theta[r], alpha,beta) %>% head(20)## # A tibble: 20 x 7
## # Groups: r [20]
## .chain .iteration .draw r theta alpha beta
## <int> <int> <int> <int> <dbl> <dbl> <dbl>
## 1 1 1 1 1 1.04 5.74 4.71
## 2 1 1 1 2 1.29 5.74 4.71
## 3 1 1 1 3 2.20 5.74 4.71
## 4 1 1 1 4 1.09 5.74 4.71
## 5 1 1 1 5 0.941 5.74 4.71
## 6 1 1 1 6 1.01 5.74 4.71
## 7 1 1 1 7 1.58 5.74 4.71
## 8 1 1 1 8 1.05 5.74 4.71
## 9 1 1 1 9 1.10 5.74 4.71
## 10 1 1 1 10 1.28 5.74 4.71
## 11 1 1 1 11 0.828 5.74 4.71
## 12 1 1 1 12 1.32 5.74 4.71
## 13 1 1 1 13 0.875 5.74 4.71
## 14 1 1 1 14 1.17 5.74 4.71
## 15 1 1 1 15 0.480 5.74 4.71
## 16 1 1 1 16 1.26 5.74 4.71
## 17 1 1 1 17 0.574 5.74 4.71
## 18 1 1 1 18 1.95 5.74 4.71
## 19 1 1 1 19 0.988 5.74 4.71
## 20 1 1 1 20 1.46 5.74 4.71chainごと、iterationごと、drawごと、地域(r)ごとのパラメータの推定結果が一つのtibble形式のデータフレームとして整理されます。パラメータが複数ある場合には、gather_draws関数を使って、変数群をlongフォーマットで整理することもできます(ggplotで可視化するときに便利です)。
事後分布の要約統計量系関数
tidybayesパッケーでは、spread_drawsもしくはgather_draws関数で整然化されたデータを下記の関数に流すことで、事後分布のようやく統計量の整然データを得ることができます。中央値、平均値、最頻値について、それぞれパーセンタイルに基づく方法と最高密度区間に基づく方法を選べます。
median_hdci: 中央値で最高密度区間mean_hdci: 平均値で最高密度区間mode_hdci: 最頻値で最高密度区間median_qi: 中央値でパーセンタイルmean_qi:平均値でパーセンタイルmode_qi:最頻値でパーセンタイル
試しに各市町村の潜在SMR\(\theta\)の事後分布の中央値と最高密度区間を抽出して見ます。
fit %>% spread_draws(theta[r]) %>%
median_hdci()## # A tibble: 59 x 7
## r theta .lower .upper .width .point .interval
## <int> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 1 0.778 0.556 0.999 0.95 median hdci
## 2 2 1.00 0.668 1.41 0.95 median hdci
## 3 3 2.22 1.71 2.78 0.95 median hdci
## 4 4 1.01 0.784 1.26 0.95 median hdci
## 5 5 1.28 0.746 1.85 0.95 median hdci
## 6 6 1.14 0.699 1.62 0.95 median hdci
## 7 7 1.06 0.546 1.64 0.95 median hdci
## 8 8 1.29 0.650 1.99 0.95 median hdci
## 9 9 1.10 0.622 1.65 0.95 median hdci
## 10 10 1.46 0.824 2.18 0.95 median hdci
## # … with 49 more rows簡単にstanオブジェクトから整然データが得られました。地域データなので、せっかくなので地図で可視化したいと思います。
library(jpndistrict)
fukushima<-jpndistrict::jpn_pref(pref_code="7")
theta_bayes<-fit %>% spread_draws(theta[r]) %>% median_hdci()
fukushima2<-data.frame(fukushima,theta_bayes,dat)
fukushima2 %>% ggplot() +
geom_sf(aes(geometry = geometry,fill=theta),col = "white" )+
theme_bw()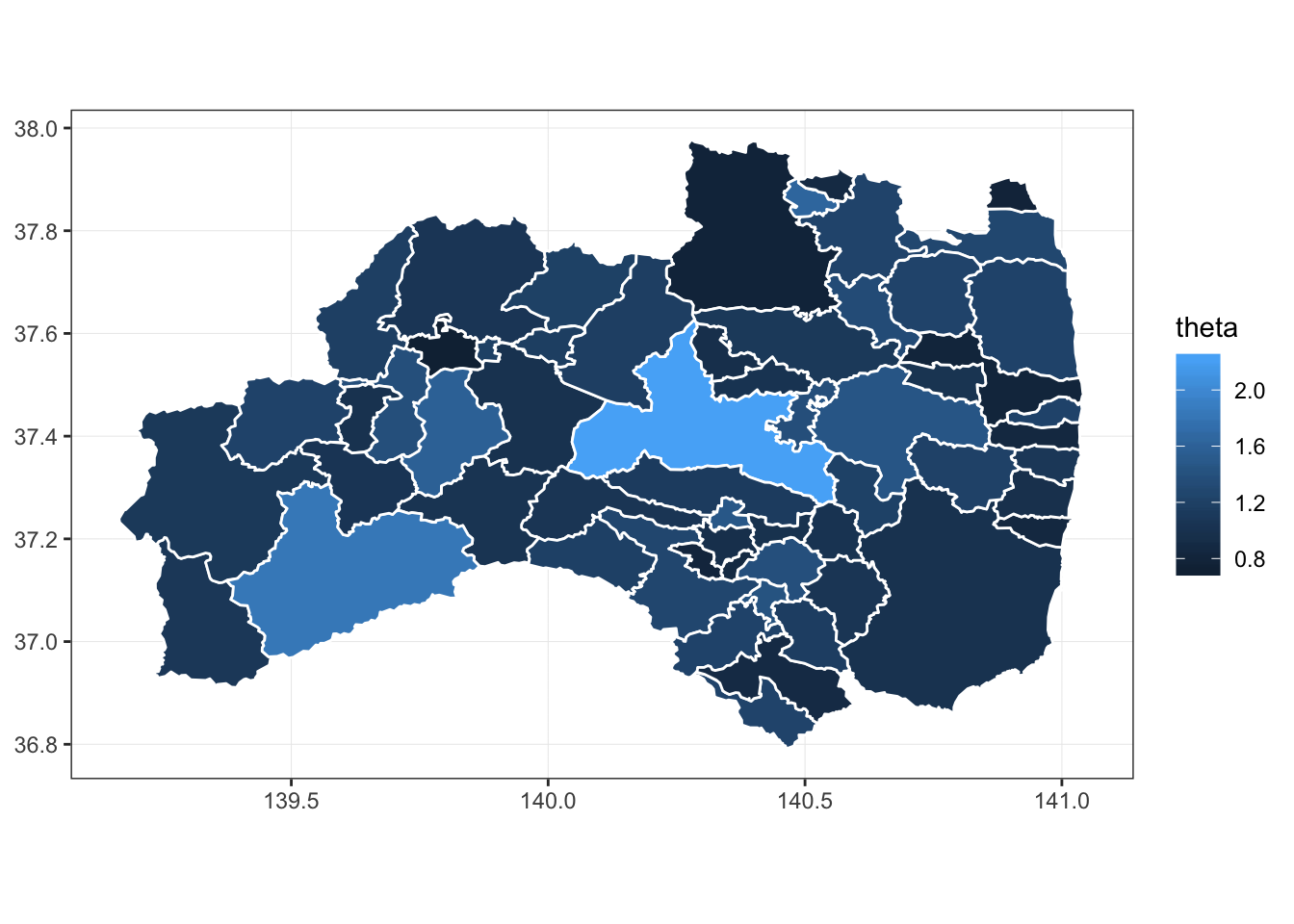
dplyrの関数がパイプ演算子でそのまま使えるので、全ての市町村から市のデータ(r=1:13)だけを抽出したい場合に、filter関数で抽出することができます。
fit %>% spread_draws(theta[r]) %>%
filter(r==grep("市",dat$ct_name))%>%
median_hdci()## # A tibble: 13 x 7
## r theta .lower .upper .width .point .interval
## <int> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 1 0.765 0.582 0.997 0.95 median hdci
## 2 2 0.992 0.673 1.36 0.95 median hdci
## 3 3 2.22 1.65 2.66 0.95 median hdci
## 4 4 0.997 0.798 1.27 0.95 median hdci
## 5 5 1.24 0.849 1.85 0.95 median hdci
## 6 6 1.15 0.710 1.63 0.95 median hdci
## 7 7 1.05 0.557 1.53 0.95 median hdci
## 8 8 1.24 0.665 2.03 0.95 median hdci
## 9 9 1.09 0.623 1.60 0.95 median hdci
## 10 10 1.50 0.884 2.11 0.95 median hdci
## 11 11 1.24 0.793 1.84 0.95 median hdci
## 12 12 1.25 0.699 1.76 0.95 median hdci
## 13 13 1.03 0.511 1.76 0.95 median hdci事後分布のプロット関数
geom_eyeh:事後分布を両側に出します。geom_harlfeyeh: 事後分布が片側だけに出ます
どちらも事後分布と共に、点推定値と確信区間が表示されます。geom_eyeh,geom_harlfeyehの引数、point_intervalで点推定値と確信区間にどれを使うか指定できます。また引数.widthで確信区間の区間幅も指定可能です。
fit %>% spread_draws(theta[r]) %>%
filter(r==grep("市",dat$ct_name)[1:2])%>% ggplot()+geom_eyeh(aes(x=theta,y=as.factor(r)),point_interval=median_hdci,.width=c(.66,0.96))+ylab("City")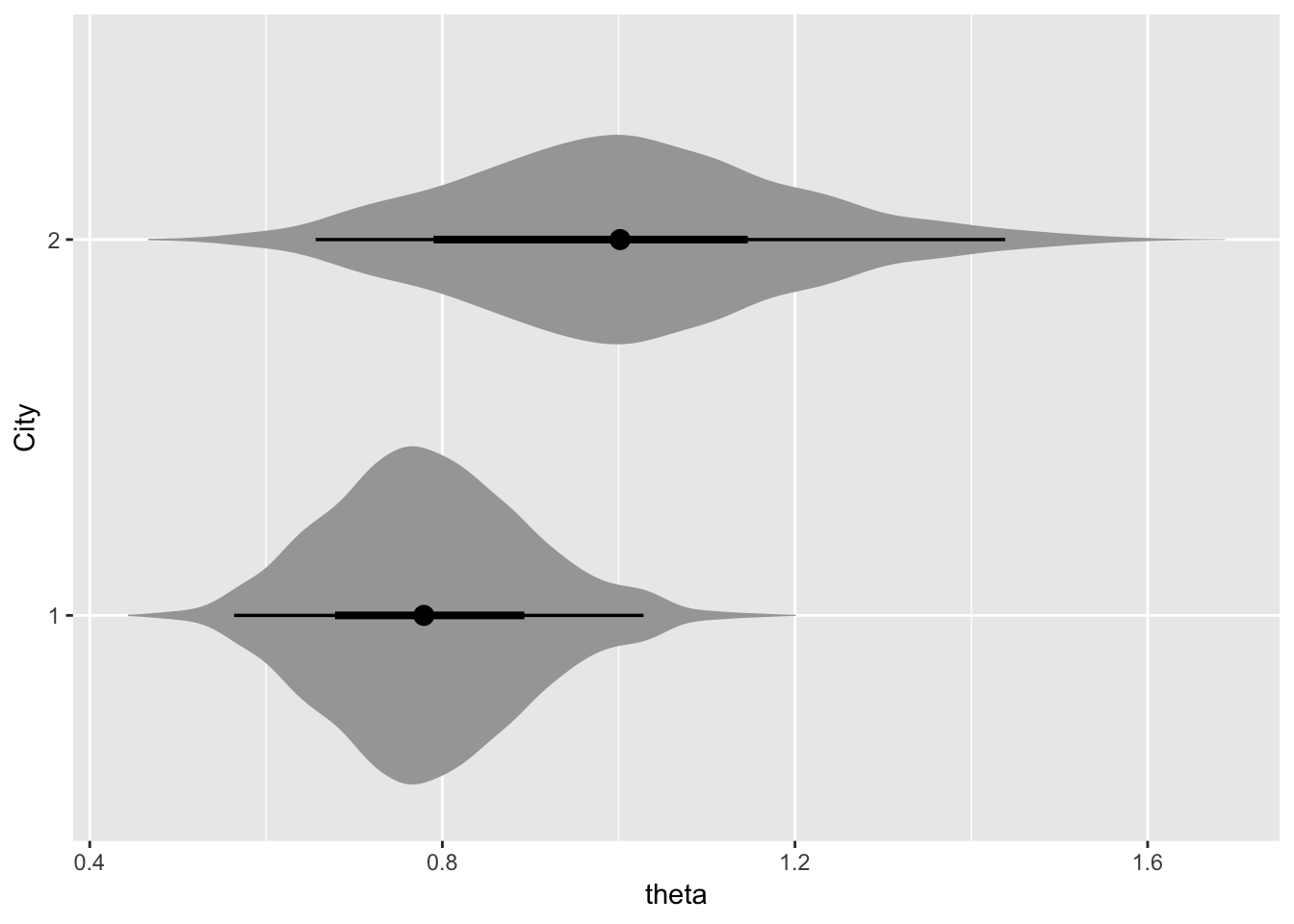
fit %>% spread_draws(theta[r]) %>%
filter(r==grep("市",dat$ct_name)[1:2])%>% ggplot()+geom_halfeyeh(aes(x=theta,y=as.factor(r)),point_interval=median_hdci,.width=c(.66,0.96))+ylab("City")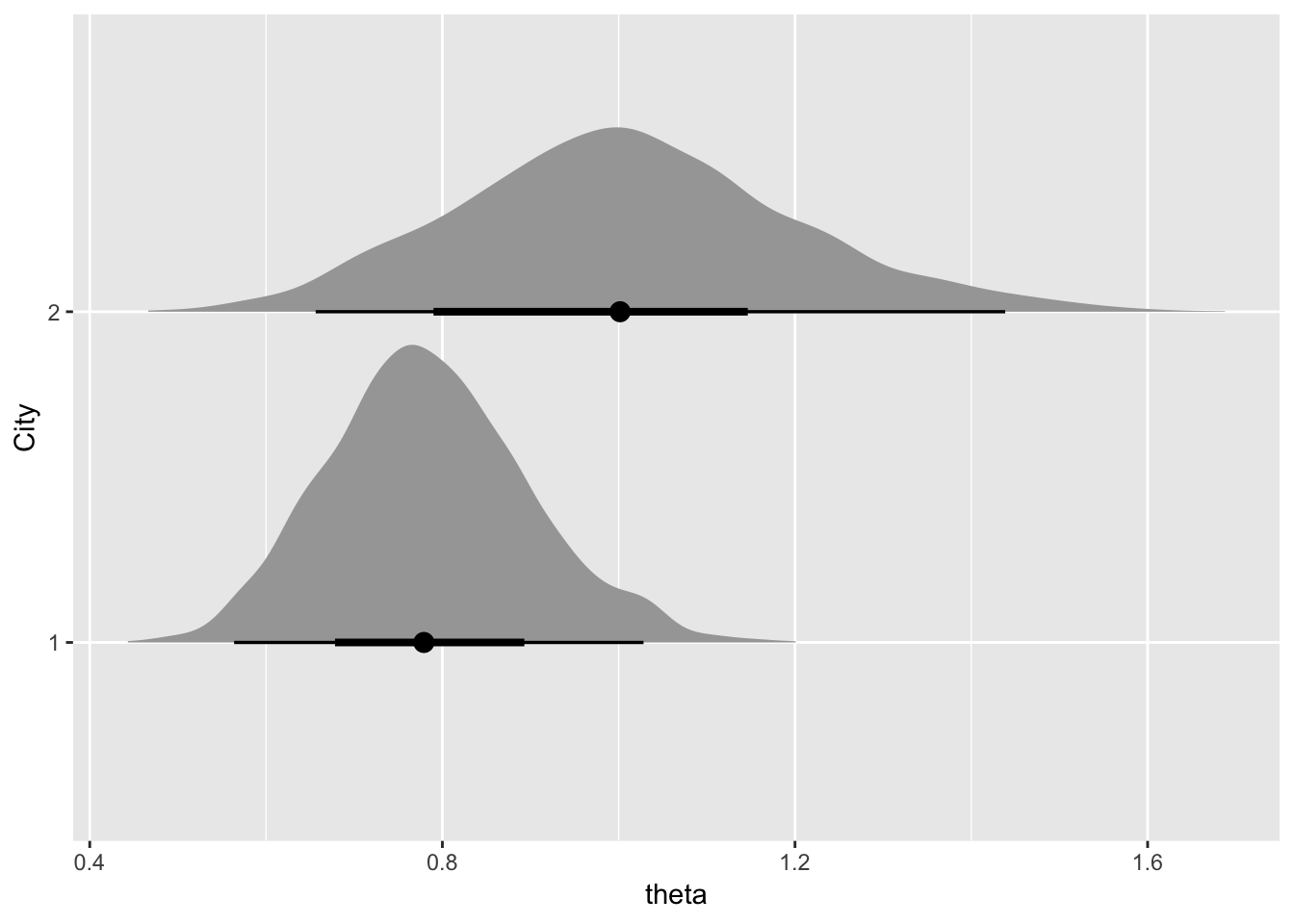
モコモコしたくない人は区間推定のみのプロットも選択可能
stat_pointintervalh:spread_drawsやspread_drawsでできた整然データをこの関数に流すことで点推定値と確信区間がプロットできます。geom_eye関数のときと同様に、引数point_intervalで点推定値と確信区間にどれを使うか指定できます。また引数.widthで確信区間の区間幅も指定可能です。
#市町村(r) 1:5までを表示
fit %>% spread_draws(theta[r]) %>%
filter(r==grep("市",dat$ct_name)[1:5])%>% ggplot()+stat_pointintervalh(aes(y = r, x = theta),point_interval=median_hdci,.width=c(.66,0.96))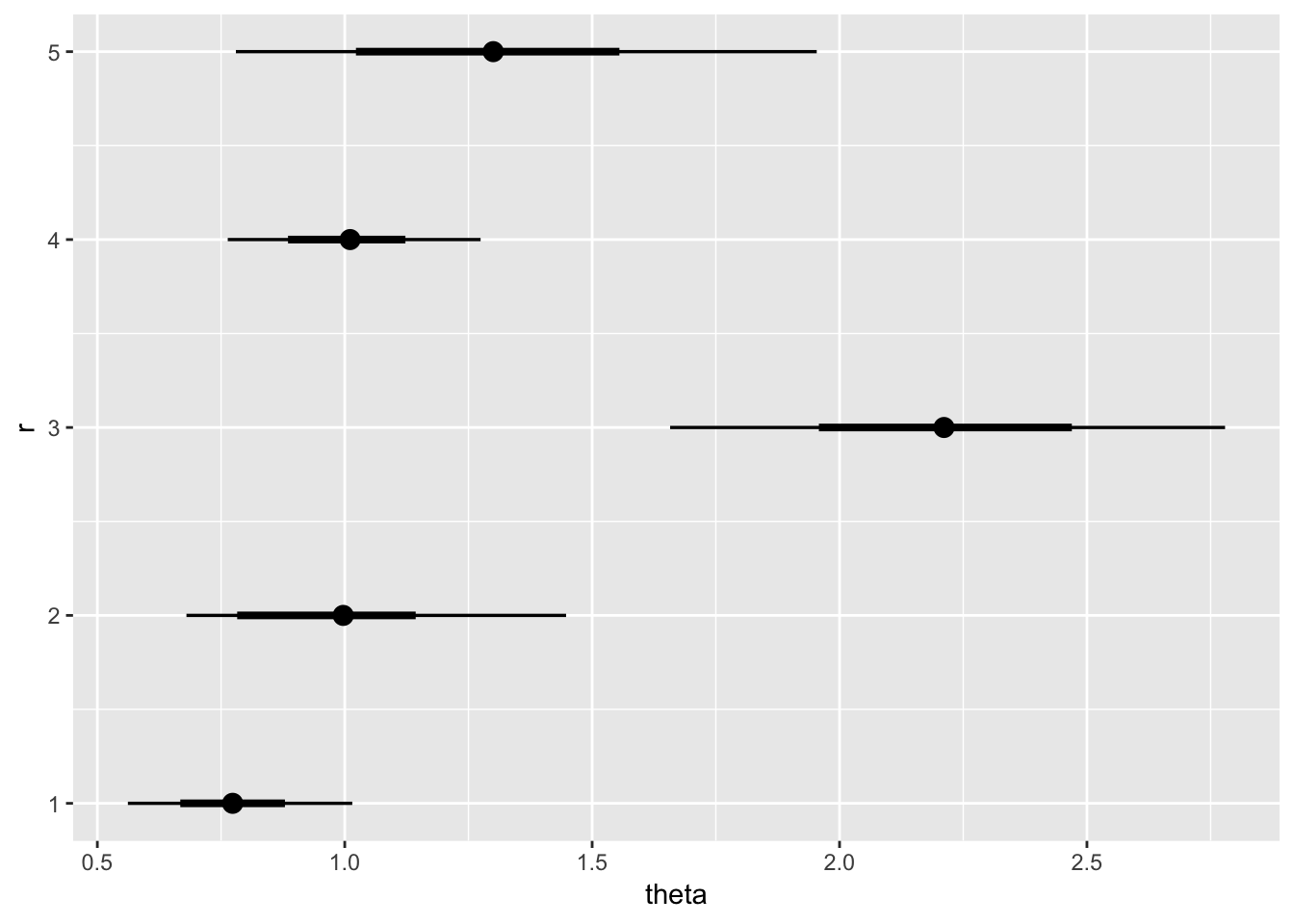
パラメータの条件間比較
compare_levels: この関数では,各条件の差の事後分布を求めてくれます。
ここでは、地域\(r\)(1から3)間のSMR(\(\theta\))の差を求めます。iterationごとに、条件間でのthetaの推定値の差を求める関数です。この結果をgeom_eyehやstat_intervalなどこれまで使い方を示してきた関数に流すと同様のプロットができます。美しい。
fit %>%
spread_draws(theta[r]) %>%
filter(r<=3) %>%
compare_levels(theta, by = r) %>%
ggplot(aes(y = r, x = theta)) +
geom_halfeyeh()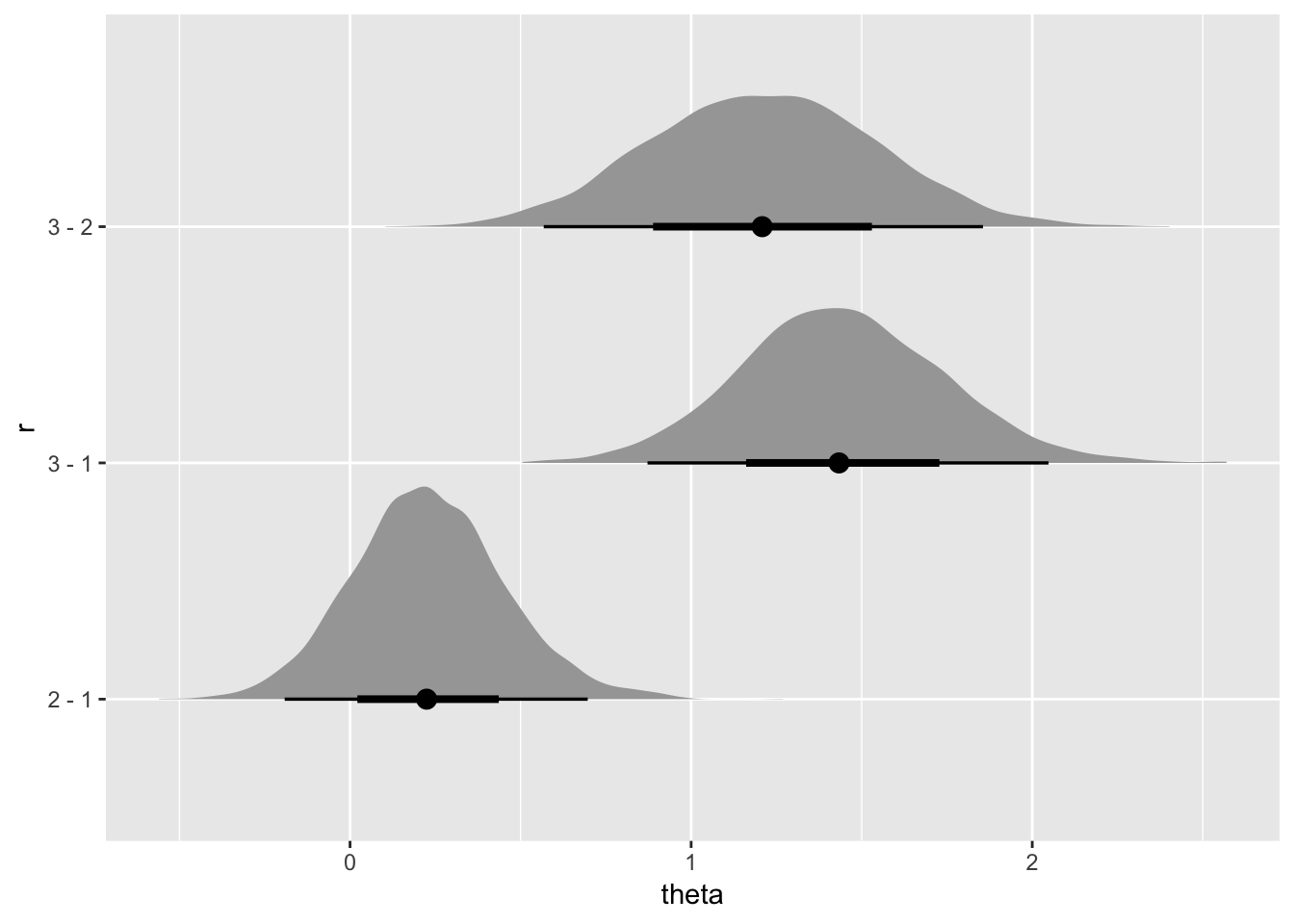
用途不明なドットプロット
かわいい以外に使い道が見出せずにいます本家のページに紹介されていたので紹介しておきます。かわいいです。
fit %>%
spread_draws(theta[r]) %>%
filter(r==grep("市",dat$ct_name)[1:2])%>% do(data_frame(theta = quantile(.$theta, ppoints(200)))) %>%
ggplot(aes(x = theta)) +
geom_dotplot(binwidth = .02) +
facet_grid(r ~ .) +
scale_y_continuous(breaks = NULL)
他のパッケージとの橋渡し
tidybayesパッケージでは、収束診断系の統計指標やプロットのための関数が用意されていません。それらはbayesplotで既にいい感じのんがあるからそっちにスムースにデータを送れるようにするのがいい、ということらしいです。
unspread_draws関数は, spread.draws関数で整然化したデータarray型に戻す関数です。これを使うと、array型のデータを読み込むbayesplotの関数にデータを送れます。
例えば、一度’spread_draws’で整然化した後に、chain1だけが振る舞いがおかしかったので、その一つだけを外して自己相関などの収束結果を確認したいというとき、下記のようにできます。
# filder 関数でchain 3だけ外したあとで、unspread_drawsでアレイ型にデータを戻してbaysplotのmcmc_acf関数にデータを流す
library(bayesplot)
fit %>%
spread_draws(theta[r]) %>% filter(.chain!=3) %>% unspread_draws(theta[r],drop_indices=T) %>% mcmc_acf(pars=c("theta[1]","theta[2]"))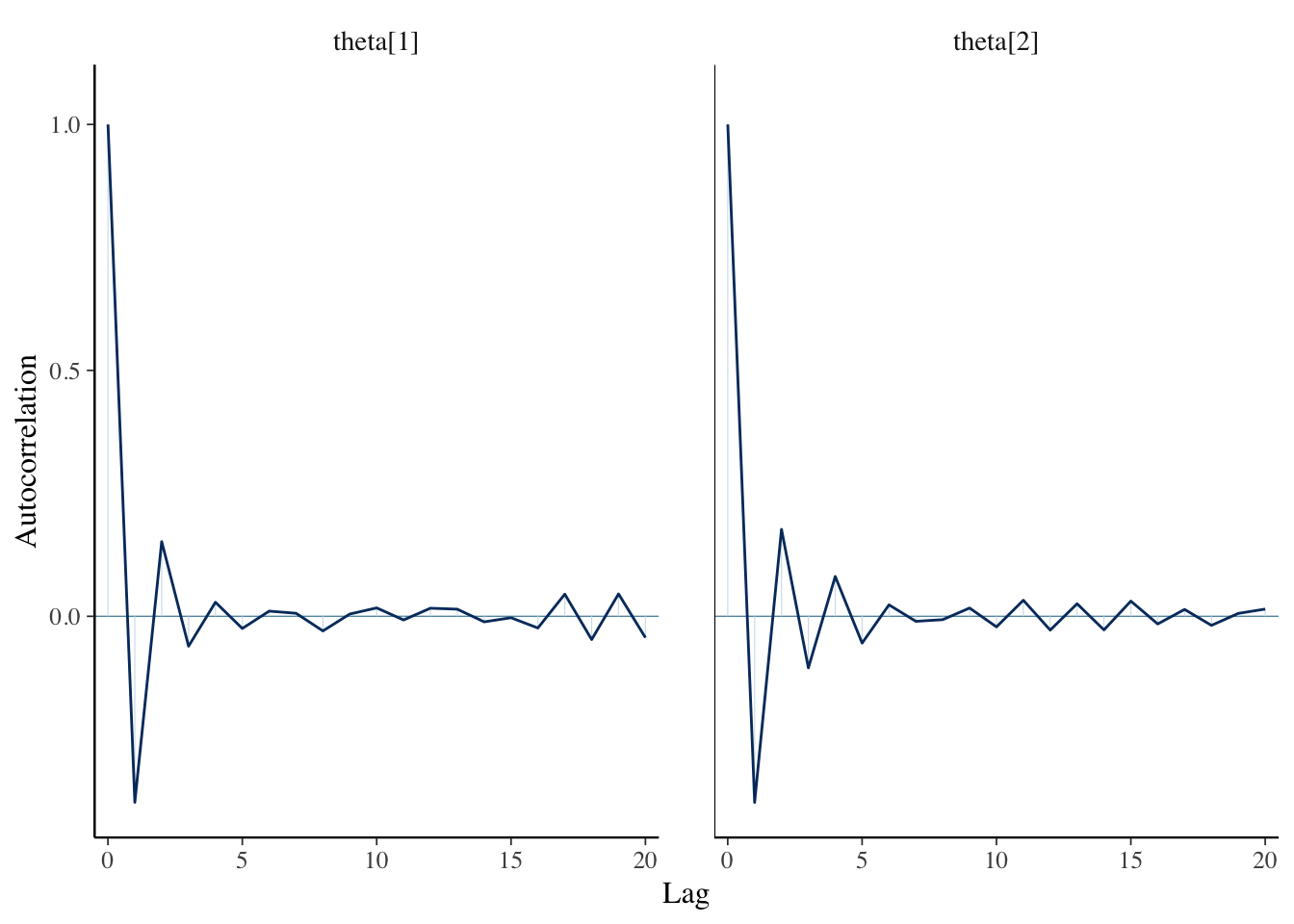
ちなみにトレースプロットを書きたかったら、spread_drawsからそのままggplotで軸を指定すればOKです。
fit %>%
spread_draws(theta[r]) %>% filter(.chain!=3,r==c(1:3)) %>%
ggplot(aes(x=.iteration,y=theta,group=as.factor(.chain),color=as.factor(.chain)))+geom_line(alpha=0.5)+facet_wrap(~r,ncol=1)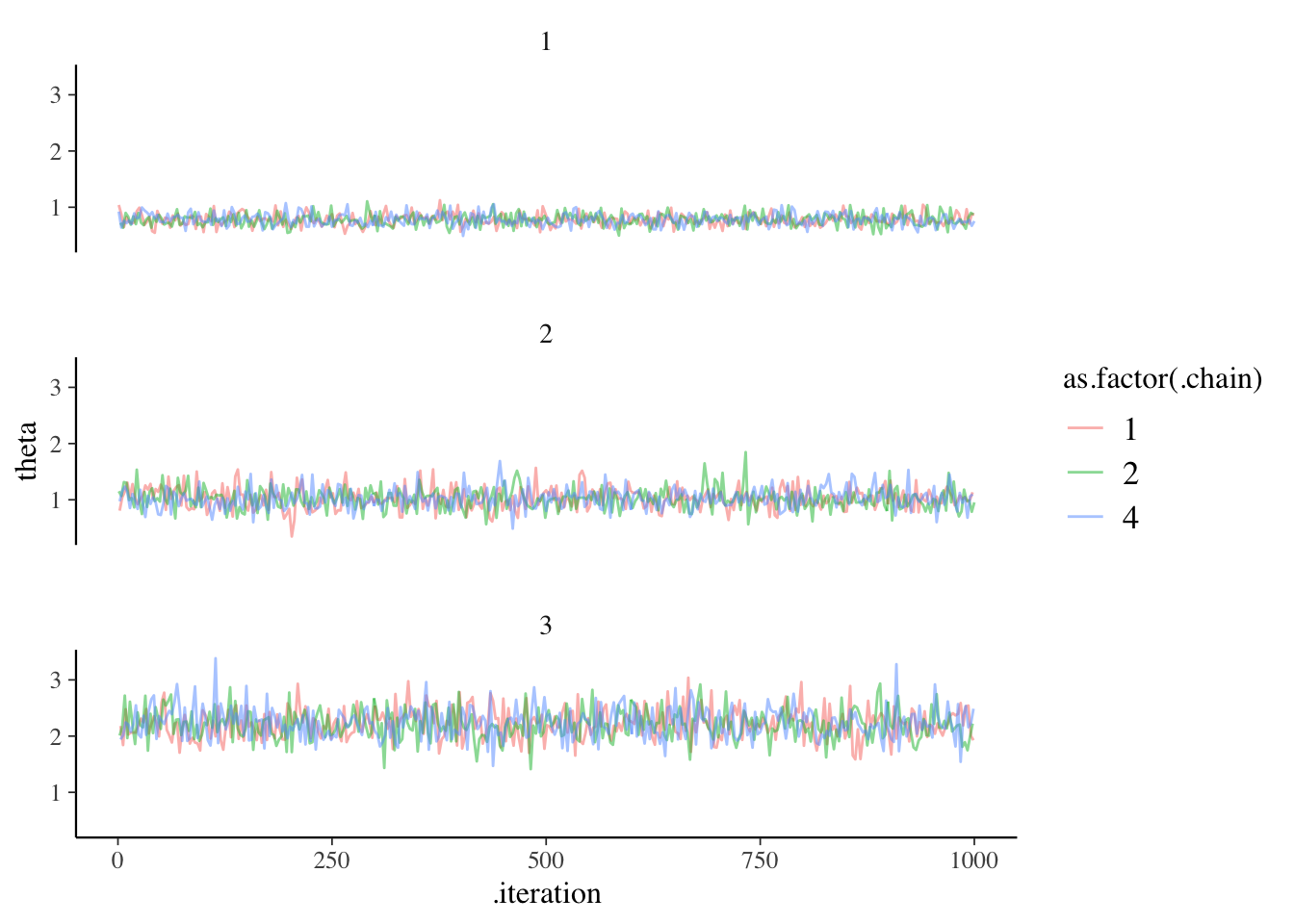
使用感
dplyrやggplotを使う人はとても使いやすいと思います。stanに内蔵されている可視化関数よりも自由度高くggplotで操作できる感覚があります。使用前ぶっちゃけあまり必要性を感じていませんでしたが、bayesplotなど他のパッケージとの橋渡し関数も周到に用意されているので、使いやすさUPです。
今回の記事では関数を全て紹介しきれていません。特に今回は、事後予測分布のプロット関数や、回帰系の予測モデルのプロットに使える関数について触れていません(魅力的な関数が盛りだくさん!!)。それらも便利そうなので、またの機会に試してみて紹介したいと思います。
Enjoy Stan with tidybayes!!